L’intelligence artificielle au service de l'imagerie orthopédique

Provient des auteurs. Licence CC
Identifier correctement des structures osseuses sur des radiographies est une étape importante pour certaines procédures en orthopédie. Cette tâche, apparemment simple, est principalement exécutée par des radiologistes avant d’effectuer des procédures plus complexes. L'identification automatique des os dans des radiographies, notamment, quand le champ de vision et la position du patient ne sont pas connus a priori et particulièrement quand il y a superposition de structures, reste une tâche ardue. L’objectif de ce travail est de pouvoir identifier automatiquement les os du membre inférieur dans des radiographies présentant différents champs de vision et deux orientations du patient. La méthode proposée utilise l’augmentation des données pour améliorer l’entraînement d’une méthode d’apprentissage profond (SegNet) permettant d’identifier les os des membres inférieurs. Nous avons obtenu, sur une base de données de validation constituée de 60 radiographies, un coefficient de Dice de 93,85 +/- 0,02 %, ce qui prouve l’utilité de la méthode proposée. Mots clés : Identification des structures osseuses, segmentation sémantique, radiographie
Une méthode d’identification des os plus performante
Plusieurs procédures orthopédiques impliquant les os des membres inférieurs nécessitent une imagerie aux rayons X du patient en position debout pour estimer correctement des paramètres cliniques (par exemple : l’angle cervico-diaphysaire, l’angle fémoro-tibial) importants pour la prise de décision. Ces paramètres cliniques sont extraits, entre autres, par l’identification d’os dans des images [1]. Des méthodes automatisées pour mettre en œuvre cette tâche [2] [3] sont décrites dans la littérature, mais la plupart d’entre elles ont été testées sur des radiographies ayant le même champ de vision et la même orientation du patient, ou sont dédiées à l’identification d’une unique structure [2] ou encore à des images relativement peu complexes (par exemple, l’analyse des images antéro-postérieures) [2].
Dans cet article, nous proposons une méthode d’identification des structures osseuses dans une radiographie présentant différents champs de vision (images de membres inférieurs, images de corps entier et images où l’os visé est partiellement visible) et deux orientations du patient (0°/90° et 45°/45°), qui ne nécessite aucune intervention humaine. La solution mise en œuvre a été appliquée à des radiographies acquises à l’aide du système EOS [4] et permet d’extraire avec précision et robustesse quatre os des membres inférieurs (fémurs et tibias) malgré des difficultés importantes liées à la superposition de structures osseuses et musculaires.
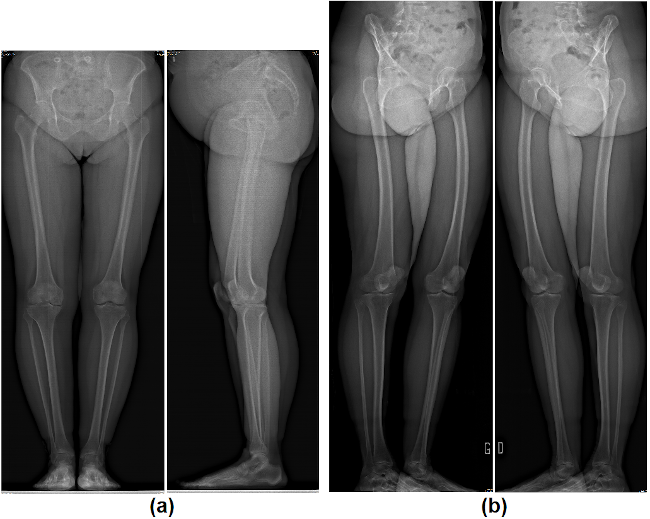
Figure 1 (a) Radiographies en orientation 0°/90° (b) Radiographies en orientation 45°/45°
Méthode proposée basée sur l’augmentation de données
Pour identifier automatiquement les quatre os, nous avons, dans un premier temps, généré des masques de vérité terrain en identifiant les contours de chaque structure osseuse sur les radiographies associées (180 au total). À l’aide de ces contours, nous créons des masques, pour chaque structure, distinguables par des couleurs différentes. Afin d’améliorer les performances d’entraînement du réseau de neurones utilisé (SegNet [5]), une stratégie d’augmentation de données a été proposée pour obtenir une base de données d’entraînement composée de 43 960 images. La stratégie d’augmentation de données est basée sur 3 transformations, à savoir : la mise en échelle (uniforme et non uniforme), le rognage et les fenêtres coulissantes. Lors de la phase d’apprentissage, le réseau prend en entrée une image et le masque associé pour apprendre les caractéristiques de la structure visée dans l’image, qui vont lui permettre d’estimer le masque. Durant la phase de test, le réseau prend en entrée une image puis génère un masque en sortie, en lien avec la structure osseuse ciblée, en utilisant les caractéristiques apprises durant la phase d’apprentissage.

Figure 2 Architecture de la méthode SegNet
Résultats de la méthode SegNet
Pour l’évaluation, nous avons utilisé une base de données constituées de 60 radiographies acquises à l’aide du système EOS. Nous avons utilisé comme métrique d’évaluation le coefficient de Dice afin de mesurer la similarité entre le masque de vérité terrain et le masque prédit. Nous avons obtenu un coefficient de Dice de 93,85 +/- 0,02 %. La figure ci-dessus présente certains des résultats qualitatifs obtenus.
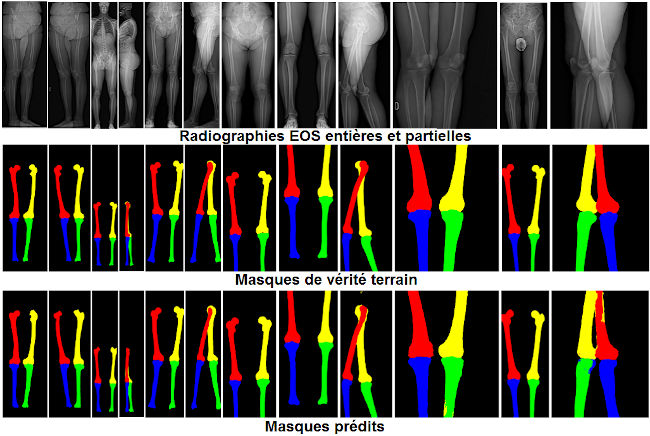
Figure 3 Comparaisons entre les masques de vérité terrain et les masques prédits par la méthode SegNet
Conclusion
Dans ce travail, quatre os des membres inférieurs sont automatiquement identifiés sur des radiographies présentant des champs de vision variables et des orientations différentes du patient. Cette solution permettra aux cliniciens de se concentrer sur des tâches beaucoup plus utiles pour le traitement personnalisé des patients.
Information supplémentaire
Pour plus d’information sur cette recherche, consulter l’article de conférence suivant :
Olory Agomma, R., Vázquez, C., Cresson, T. et de Guise, J., 2019. « Detection and Identification of Lower-Limb Bones in biplanar X-Ray Images with Arbitrary Field of View and Various Patient Orientations ». In 2019 IEEE 16th International Symposium on Biomedical Imaging (ISBI 2019).



