Une méthode simple de segmentation faiblement supervisée
Achetée sur Istock.com. Droits d’auteur.
Segmentation sémantique d’images
La segmentation sémantique d’images, qui consiste à dessiner les contours d’un objet dans une image, est un sujet de recherche intense et très utile à l’imagerie médicale. Non seulement aide-t-elle à lire et à comprendre les tomographies, mais elle sert également au diagnostic, à la planification d’opérations et au suivi de nombreuses maladies; elle représente un potentiel énorme en médecine personnalisée.
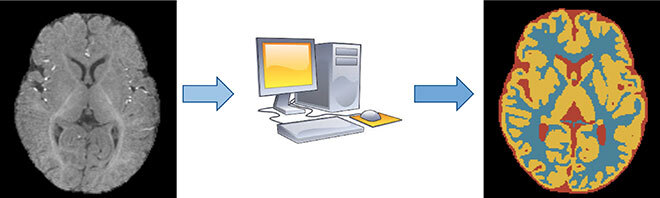
Ces dernières années, les réseaux neuronaux convolutifs profonds (CNN) ont dominé dans le domaine de la segmentation sémantique, tant en vision par ordinateur qu’en imagerie médicale; ils réalisent des performances révolutionnaires, mais au prix d’énormes quantités de données annotées. Un ensemble de données est un groupe d’exemples, chacun contenant à la fois une image et sa segmentation correspondante (c’est-à-dire la vérité terrain), comme le montre la figure 1. Grâce à des méthodes d’optimisation comme l’algorithme du gradient stochastique, le réseau est entraîné jusqu’à ce que ses données de sortie correspondent le plus possible à la vérité terrain. Une fois le réseau entraîné, il peut servir à prédire la segmentation sur de nouvelles images inédites.
Étiqueter les données est coûteux
Dans le domaine médical, seuls les médecins formés peuvent annoter en toute confiance les images pour la segmentation, un processus très coûteux. En outre, la complexité et la taille des images peuvent ralentir l’annotation, nécessitant plusieurs jours pour les scans cérébraux complexes. Il s’avère donc nécessaire de trouver des méthodes plus efficace.
Un axe de recherche attrayant consiste à faire des annotations incomplètes, ou faibles, au lieu d’annotations complètes de pixels. Dessiner un point unique à l’intérieur d’un objet est infiniment plus rapide et plus simple que d’en dessiner le contour exact. Mais il n’est pas possible d’utiliser ces annotations directement pour former un réseau de neurones, car le réseau n’a pas assez d’information pour trouver une solution optimale, voire satisfaisante.
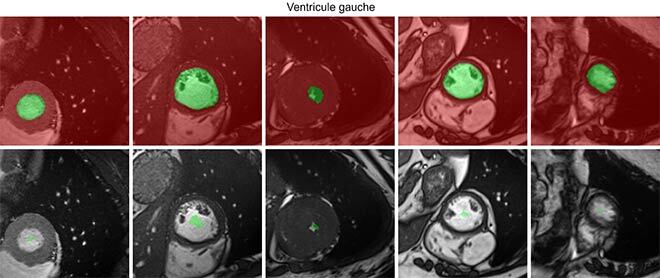
Figure 1 : Exemples d’annotations : complètes et faibles. En rouge, les pixels étiquetés arrière-plan, et en vert les pixels étiquetés avant-plan : ce qui doit être segmenté. L’absence de couleur (dans les annotations faibles) signifie qu’aucune information n’est disponible sur ces pixels pour l’apprentissage.
Notre contribution : forcer le système à considérer la taille en appliquant des pénalités
Néanmoins, il existe d’autres informations, mais elles ne figurent pas sous forme d’annotations directes. Par exemple, nous connaissons les propriétés anatomiques des organes que nous voulons segmenter, comme la taille approximative :
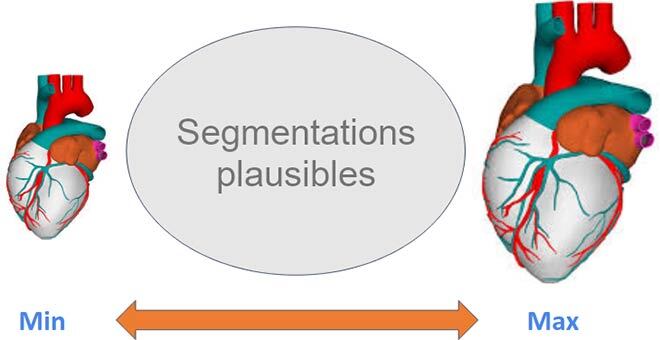
Figure 2 : En connaissant la taille approximative, nous pouvons guider le réseau vers des solutions anatomiquement réalisables
Nous pouvons donc corriger le processus d’entraînement, en forçant le réseau non seulement à prédire correctement les quelques pixels étiquetés, mais aussi à s’assurer que la taille de la segmentation prédite figure parmi les solutions anatomiquement plausibles connues au préalable. Il s’agit d’un problème d’optimisation sous contraintes, un domaine bien étudié en général (par les méthodes lagrangiennes), mais rarement appliqué aux réseaux de neurones. Le grand nombre de paramètres du réseau et des ensembles de données, et la nécessité d’alterner entre différents modes d’entraînement rendent cette solution impraticable.
Notre principale contribution a été d’inclure ces contraintes à l’étape de l’entraînement en appliquant tout simplement une pénalité directe. Ainsi, le réseau est pénalisé chaque fois que la taille de la segmentation prévue est hors limites (plus l’erreur est grande, plus la pénalité est importante), et ne l’est pas quand les limites sont respectées.
Résultats et conclusion
Étonnamment, et bien que contraire à tout manuel sur l’optimisation sous contraintes, notre simple pénalité pour apprentissage profond s’est avérée beaucoup plus efficace, rapide et stable que les méthodes lagrangiennes plus complexes. Tenir compte des informations sur la taille pendant l’entraînement, soit essentiellement ajouter un seul paramètre, nous a permis de presque combler l’écart entre supervision complète et supervision faible dans la segmentation du ventricule gauche, en annotant seulement 0,01 % des pixels.
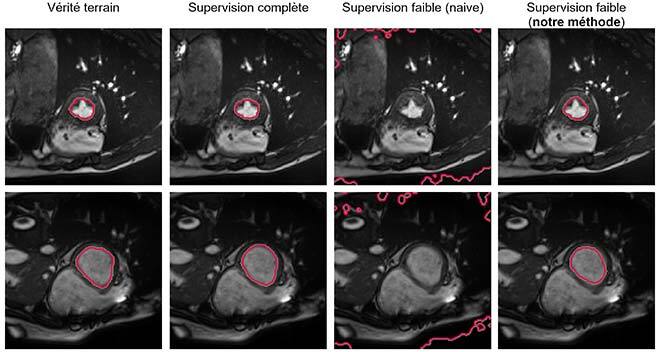
Figure 3 : Comparaison visuelle des résultats. Dans l’ordre habituel : étiquette, apprentissage avec annotations complètes, apprentissage avec simples annotations partielles, notre méthode.

Informations supplémentaires
Voir l’article complet pour des détails plus techniques décrivant les différents paramètres de plusieurs ensembles de données : Kervadec, H. ; Dolz, J. ; Tang M. ; Granger, E. Boykov, Y.; Ben Ayed I. 2019. « Constrained-CNN losses for weakly supervised segmentation » Medical Image Analysis. Volume 54. pp. 88-99.
Le code, en ligne, est libre d’utilisation et de modifications : https://github.com/liviaets/sizeloss_wss.



